dplyr 팩키지로 데이터프레임 솜씨있게 조작
Overview
Teaching: 40 min
Exercises: 15 minQuestions
본인이 반복하지 않고 데이터프레임을 어떻게 하면 솜씨있게 조작할 수 있을까?
Objectives
dplyr팩키지를 파이프와 연결하여 데이터프레임을 조작하는데 6개 ‘동사’를 사용한다.데이터셋을 요약하는데
group_by()와summarize()함수를 조합하는 방법을 이해한다.논리 필터링 연산을 사용해서 데이터 일부를 분석할 수 있다.
데이터프레임을 솜씨있게 조작하는 것은 많은 과학연구원에게 많은 것을 의미한다. 특정 관측점(행) 혹은 변수(열)을 선택하거나, 특정 변수(들)로 데이터를 집단으로 그룹짓거나, 요약 통계량을 계산하기도 한다. 이런 연산작업에 정규 기본 베이스(Base) R 연산을 사용한다:
mean(gapminder[gapminder$continent == "Africa", "gdpPercap"])
[1] 2193.755
mean(gapminder[gapminder$continent == "Americas", "gdpPercap"])
[1] 7136.11
mean(gapminder[gapminder$continent == "Asia", "gdpPercap"])
[1] 7902.15
하지만, 그다지 멋있지는 않은데, 이유는 상당한 반복이 상존하기 때문이다. 여러분이 직접 반복하게 되면, 일단 여러분 시간을 지금 물론이고, 나중에 소중한 시간을 까먹게 되고, 더나아가 잠재적으로 버그가 스며들 여지를 남기게 된다.
dplyr 팩키지
운좋게도, dplyr 팩키지가
데이터프레임을 솜씨있게 조작하는데 있어 유용한 함수를 많이 제공한다.
이를 통해서, 위에서 언급된 반복을 줄이고, 실수를 범할 확률도 줄이고,
심지어 타이핑하는 수고도 줄일 수 있다.
보너스로, dplyr 문법은 훨씬 더 가독성도 높다.
가장 흔히 사용되는 6가지 함수 뿐만 아니라,
이런 함수를 조합하는데 사용되는 파이프 (%>%) 연산자 사용법도 다룬다.
select()filter()group_by()summarize()mutate()
이전 수업에서 팩키지를 설치하지 않았다면, 설치해서 직접 실습해보기 바란다:
install.packages('dplyr')
이제 팩키지를 불러와서 적재한다:
library("dplyr")
select() 사용
예를 들어, 데이터프레임에서 변수 일부만 뽑아서 작업해 나가고자 한다면,
select() 함수를 사용한다.
이 함수는 선택한 변수만 갖도록 지정한다.
year_country_gdp <- select(gapminder,year,country,gdpPercap)
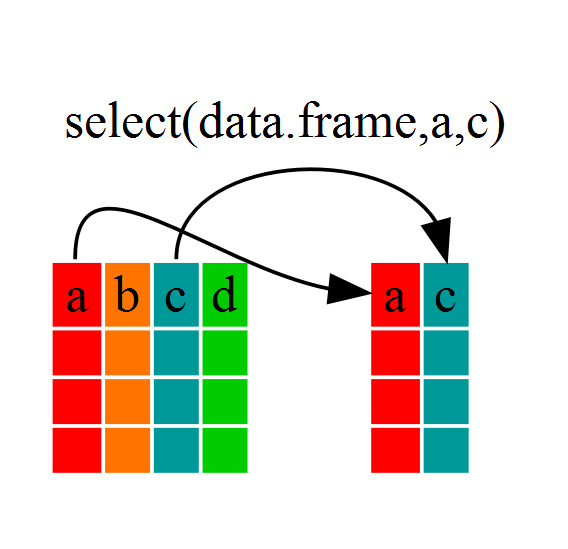
year_country_gdp 데이터프레임을 열게되면,
year, country, gdpPercap 변수만 담겨있는 것을 보게 된다.
위에서는 정규 문법이 사용되었지만,
dplyr 팩키지의 장점은 파이프를 사용해서 함수 다수를 조합하는데 있다.
파이프 문법은 이전에 R에서 살펴봤던 것과는 사뭇 다르다.
위에서 파이프를 사용했던 것을 다시 작성해본다.
year_country_gdp <- gapminder %>% select(year,country,gdpPercap)
파이프를 사용해서 작성한 이유에 대한 이해를 돕기 위해서, 단계별로 살펴보자.
먼저 gapminder 데이터프레임을 불러오고 나서,
%>% 파이프 기호를 사용해서 다음 작업단계(select() 함수)로 전달했다.
이번 경우에는 select() 함수에 데이터 객체를 명세하지 않았다.
이유는 이전 파이프로부터 건네받았기 때문이다.
재미난 사실: 유닉스 쉘 강의에서 이미 파이프를 접해봤을 것이다.
R에서 파이프 기호가 %>%인 반면, 쉘에서는 |을 사용한다.
하지만, 개념은 동일하다!
filter() 사용하기
이제 앞선 작업을 바탕으로 앞으로 작업을 진척시켜 보자.
유럽대륙만 갖고 작업하고자 한다. select 와 filter를 조합하면 된다.
year_country_gdp_euro <- gapminder %>%
filter(continent=="Europe") %>%
select(year,country,gdpPercap)
도전과제 1
명령어를 하나 (여러 행에 걸칠 수 있고, 파이프도 포함한다) 작성하는데,
lifeExp,country,year변수에 대해서 아프리카 대륙(African)만 갖는 데이터프레임을 작성한다. 하지만, 다른 대륙은 포함되면 안된다. 데이터프레임 행의 갯수는 얼마나 되는가? 그리고 이유는 무엇인가?도전과제 1에 대한 해답
year_country_lifeExp_Africa <- gapminder %>% filter(continent=="Africa") %>% select(year,country,lifeExp)
지난번과 마찬가지로, gapminder 데이터프레임을 filter() 함수에 전달하고 나서,
필터링된 gapminder 데이터프레임 버젼을 select() 함수에 전달한다.
주의: 연산순서가 이번 경우에 무척 중요하다.
select() 함수를 먼저 실행하면, filter() 함수는 대륙 변수를 찾을 수 없는데,
이유는 이전 단계에서 제거했기 때문이다.
group_by()와 summarize() 사용하기
이제, 기본 베이스(base) R로 작업함으로써 실수를 범하기 쉬운 반복작업을 줄일 것으로 생각했지만,
현재까지 목표를 달성하지 못했다. 왜냐하면, 각 대륙마다 상기 작업을 반복해야 되기 때문이다.
filter() 대신에, group_by()를 사용한다.
filter()는 특정 기준을 만족하는 관측점만 넘겨준다(이번 경우: continent=="Europe").
group_by()는 본질적으로, 필터에서 사용할 수 있는 모든 유일무이한 기준을 사용할 수 있다.
str(gapminder)
'data.frame': 1704 obs. of 6 variables:
$ country : Factor w/ 142 levels "Afghanistan",..: 1 1 1 1 1 1 1 1 1 1 ...
$ year : int 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num 8425333 9240934 10267083 11537966 13079460 ...
$ continent: Factor w/ 5 levels "Africa","Americas",..: 3 3 3 3 3 3 3 3 3 3 ...
$ lifeExp : num 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num 779 821 853 836 740 ...
str(gapminder %>% group_by(continent))
Classes 'grouped_df', 'tbl_df', 'tbl' and 'data.frame': 1704 obs. of 6 variables:
$ country : Factor w/ 142 levels "Afghanistan",..: 1 1 1 1 1 1 1 1 1 1 ...
$ year : int 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num 8425333 9240934 10267083 11537966 13079460 ...
$ continent: Factor w/ 5 levels "Africa","Americas",..: 3 3 3 3 3 3 3 3 3 3 ...
$ lifeExp : num 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num 779 821 853 836 740 ...
- attr(*, "vars")= chr "continent"
- attr(*, "drop")= logi TRUE
- attr(*, "indices")=List of 5
..$ : int 24 25 26 27 28 29 30 31 32 33 ...
..$ : int 48 49 50 51 52 53 54 55 56 57 ...
..$ : int 0 1 2 3 4 5 6 7 8 9 ...
..$ : int 12 13 14 15 16 17 18 19 20 21 ...
..$ : int 60 61 62 63 64 65 66 67 68 69 ...
- attr(*, "group_sizes")= int 624 300 396 360 24
- attr(*, "biggest_group_size")= int 624
- attr(*, "labels")='data.frame': 5 obs. of 1 variable:
..$ continent: Factor w/ 5 levels "Africa","Americas",..: 1 2 3 4 5
..- attr(*, "vars")= chr "continent"
..- attr(*, "drop")= logi TRUE
group_by() 함수를 사용한 데이터프레임 구조(grouped_df)가 원래 gapminder 데이터프레임 구조(data.frame)와 같지 않음에 주목한다.
grouped_df는 list 리스트로 간주될 있는데, list에 각 항목은 data.frame으로,
각 데이터프레임은 특정 대륙 continent에 대응되는 행만 담겨진다(적어도 상기 예제의 경우).
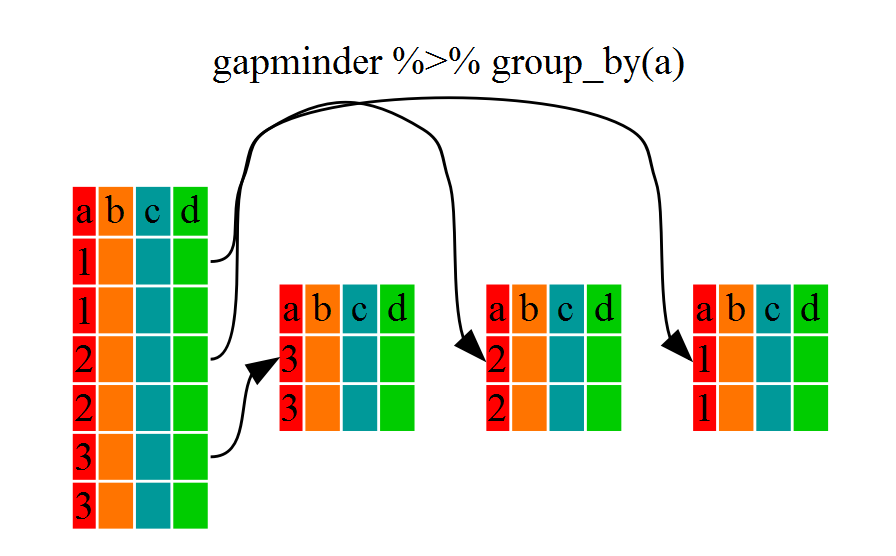
summarize() 사용하기
상기 예제는 그다지 특별한 점이 없다.
왜냐하면 group_by() 함수는 summarize()와 함께할 때 훨씬 더 흥미롭다.
두 함수를 조합하면 새로운 변수가 생성되는데,
각 대륙별 데이터프레임에 대해 반복적인 함수 작업을 수행할 수 있다.
다시 말해, group_by() 함수를 사용해서,
최초 데이터프레임을 다수 조각으로 쪼개고 나서,
각각에 대해 함수(예를 들어 mean() 혹은 sd())를 summarize() 내부에서 실행시키게 된다.
gdp_bycontinents <- gapminder %>%
group_by(continent) %>%
summarize(mean_gdpPercap=mean(gdpPercap))
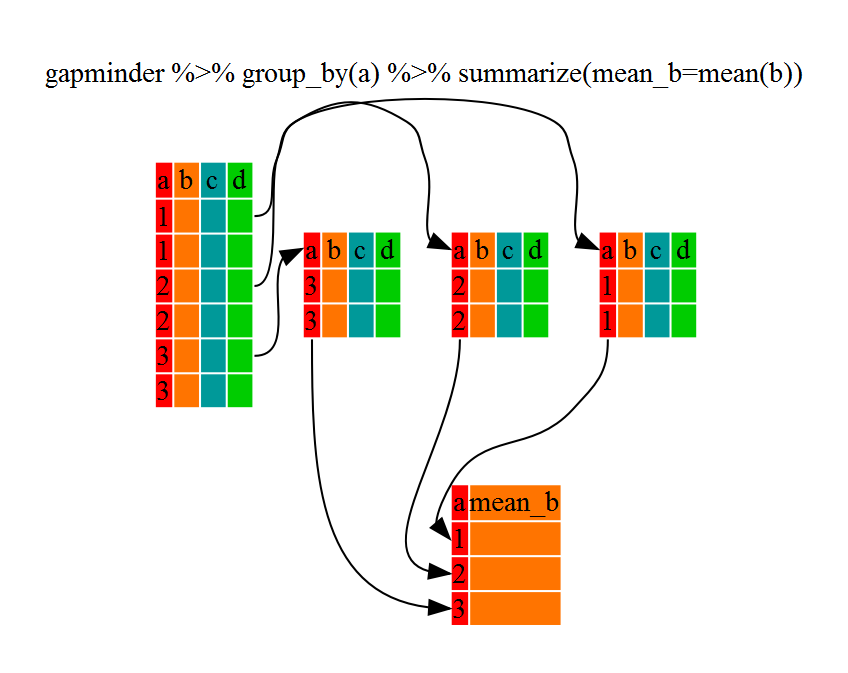
continent mean_gdpPercap
<fctr> <dbl>
1 Africa 2193.755
2 Americas 7136.110
3 Asia 7902.150
4 Europe 14469.476
5 Oceania 18621.609
상기 코드는 각 대륙별로 평균 gdpPercap를 계산할 수 있게 하지만, 훨씬 더 낫다.
도전과제 2
국가별로 평균 기대수명을 계산한다. 어느 국가가 가장 평균 기대수명이 길고, 어느 국가가 가장 평균 기대수명이 짧은가?
도전과제 2에 대한 해답
lifeExp_bycountry <- gapminder %>% group_by(country) %>% summarize(mean_lifeExp=mean(lifeExp)) lifeExp_bycountry %>% filter(mean_lifeExp == min(mean_lifeExp) | mean_lifeExp == max(mean_lifeExp))# A tibble: 2 x 2 country mean_lifeExp <fct> <dbl> 1 Iceland 76.5 2 Sierra Leone 36.8문제를 프는 또 다른 방식은
dplyr팩키지arrange()함수를 사용하는 것이다.arrange()함수는 변수에 따라 데이터프레임을 행으로 정렬하는 기능을 갖고 있다.dplyr팩키지 다른 함수처럼 유사한 구문을 갖추고 있다.arrange()함수 내부에desc()함수를 사용해서 내림차순으로 정렬할 수 있다.lifeExp_bycountry %>% arrange(mean_lifeExp) %>% head(1)# A tibble: 1 x 2 country mean_lifeExp <fct> <dbl> 1 Sierra Leone 36.8lifeExp_bycountry %>% arrange(desc(mean_lifeExp)) %>% head(1)# A tibble: 1 x 2 country mean_lifeExp <fct> <dbl> 1 Iceland 76.5
group_by() 훔수에 변수 다수를 사용해서 집단으로 그룹을 만들 수도 있다.
year 와 continent 변수로 그룹을 만들어 보자.
gdp_bycontinents_byyear <- gapminder %>%
group_by(continent,year) %>%
summarize(mean_gdpPercap=mean(gdpPercap))
이미 매우 막강한 기능이지만, 더 좋게 만들 수 있다!
summarize() 함수에 변수 하나를 정의하는 것에 한정되지 않고, 확장도 가능하다.
gdp_pop_bycontinents_byyear <- gapminder %>%
group_by(continent,year) %>%
summarize(mean_gdpPercap=mean(gdpPercap),
sd_gdpPercap=sd(gdpPercap),
mean_pop=mean(pop),
sd_pop=sd(pop))
count()와 n()
매우 흔한 연산 중 하나는 각 그룹마다 관측점 수를 세는 것이다.
dplyr 팩키지에 개수를 돕는 연관 함수가 2개 있다.
예를 들어, 2002년 데이터셋에 포함된 국가수를 확인하고자 한다면,
count() 함수를 사용하는데, 관심있는 그룹을 포함하는 칼럼을 하나이상 지정할 수도 있다.
선택사항으로 sort=TRUE를 인자로 추가하면 내림차순으로 결과를 정렬할 수 있다:
gapminder %>%
filter(year == 2002) %>%
count(continent, sort = TRUE)
# A tibble: 5 x 2
continent n
<fct> <int>
1 Africa 52
2 Asia 33
3 Europe 30
4 Americas 25
5 Oceania 2
계산과정에서 관측점 갯수를 파악할 필요가 있는 경우, n() 함수가 유용하다.
예를 들어, 각 대륙별 기대수명 표준오차를 다음과 같이 구할 수도 있다:
gapminder %>%
group_by(continent) %>%
summarize(se_le = sd(lifeExp)/sqrt(n()))
# A tibble: 5 x 2
continent se_le
<fct> <dbl>
1 Africa 0.366
2 Americas 0.540
3 Asia 0.596
4 Europe 0.286
5 Oceania 0.775
요약 연산을 몇개 엮어서 계산할 수도 있다;
이 경우 각 대륙별 기대수명에 대한 minimum, maximum, mean, se 값을 다음과 같이 계산한다:
gapminder %>%
group_by(continent) %>%
summarize(
mean_le = mean(lifeExp),
min_le = min(lifeExp),
max_le = max(lifeExp),
se_le = sd(lifeExp)/sqrt(n()))
# A tibble: 5 x 5
continent mean_le min_le max_le se_le
<fct> <dbl> <dbl> <dbl> <dbl>
1 Africa 48.9 23.6 76.4 0.366
2 Americas 64.7 37.6 80.7 0.540
3 Asia 60.1 28.8 82.6 0.596
4 Europe 71.9 43.6 81.8 0.286
5 Oceania 74.3 69.1 81.2 0.775
mutate() 사용하기
mutate() 함수를 사용해서 정보를 요약하기 전에(혹은 후에도) 새로운 변수를 생성할 수 있다.
gdp_pop_bycontinents_byyear <- gapminder %>%
mutate(gdp_billion=gdpPercap*pop/10^9) %>%
group_by(continent,year) %>%
summarize(mean_gdpPercap=mean(gdpPercap),
sd_gdpPercap=sd(gdpPercap),
mean_pop=mean(pop),
sd_pop=sd(pop),
mean_gdp_billion=mean(gdp_billion),
sd_gdp_billion=sd(gdp_billion))
mutate() 함수를 논리 필터와 연결하기: ifelse
변수를 새로 만들 때, mutate() 함수를 논리 조건문과 엮을 수 있다.
mutate() 와 ifelse()의 단순 조합을 통해서 필요한 것만 적절히 필터링할 수 있다: 즉, 새로운 무언가를 생성하는 순간에 말이다.
이러한 가독성 높은 문장을 통해서 (데이터프레임 전체 차원을 변경시키지 않고도) 특정 데이터를 버리거나,
조건에 따라 값을 갱신하는데 신속하고 강력한 방식을 제공할 수 있게 된다.
## keeping all data but "filtering" after a certain condition
# calculate GDP only for people with a life expectation above 25
gdp_pop_bycontinents_byyear_above25 <- gapminder %>%
mutate(gdp_billion = ifelse(lifeExp > 25, gdpPercap * pop / 10^9, NA)) %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
sd_gdpPercap = sd(gdpPercap),
mean_pop = mean(pop),
sd_pop = sd(pop),
mean_gdp_billion = mean(gdp_billion),
sd_gdp_billion = sd(gdp_billion))
## updating only if certain condition is fullfilled
# for life expectations above 40 years, the gpd to be expected in the future is scaled
gdp_future_bycontinents_byyear_high_lifeExp <- gapminder %>%
mutate(gdp_futureExpectation = ifelse(lifeExp > 40, gdpPercap * 1.5, gdpPercap)) %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
mean_gdpPercap_expected = mean(gdp_futureExpectation))
dplyr 을 ggplot2와 조합하기
ggplot2을 사용해서 퍼싯(facet) 패널 계층을 추가해서 작은 창에 그래프를 담아내는 방식을
앞선 시각화 수업에서 확인했따. 다음에 앞서 사용한 코드가 나와 있다:
# Get the start letter of each country
starts.with <- substr(gapminder$country, start = 1, stop = 1)
# Filter countries that start with "A" or "Z"
az.countries <- gapminder[starts.with %in% c("A", "Z"), ]
# Make the plot
ggplot(data = az.countries, aes(x = year, y = lifeExp, color = continent)) +
geom_line() + facet_wrap( ~ country)
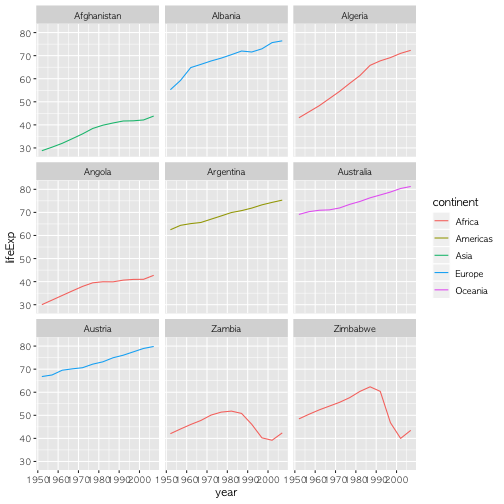
상기 코드는 원하는 그래프를 만들어 주지만,
다른 용도도 뚜렷이 없는 변수(starts.with, az.countries)도 생성시키게 된다.
%>% 연산자를 사용해서 dplyr 함수를 엮어 데이터를 파이프에 흘러 보냈듯이,
ggplot() 함수에 데이터를 흘러 보낼 수 있다.
%>% 파이프 연산자가 함수의 첫번째 인자를 대체하기 때문에,
ggplot() 함수에 data = 인자를 명세할 필요는 없다.
dplyr, ggplot2 함수를 조합하게 되면, 동일한 그래프를 생성하는데 있어
변수를 새로 생성시키거나 데이터를 변경할 필요가 없어진다.
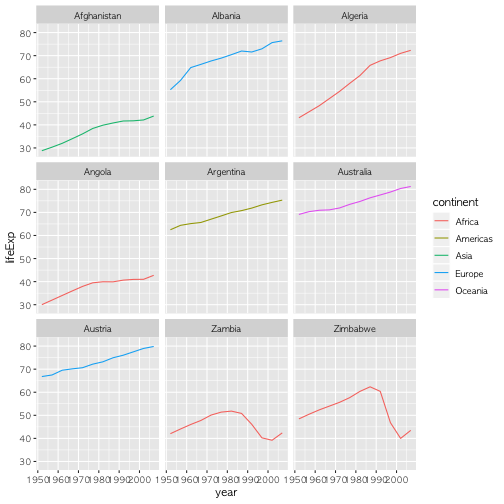
gapminder %>%
# Get the start letter of each country
mutate(startsWith = substr(country, start = 1, stop = 1)) %>%
# Filter countries that start with "A" or "Z"
filter(startsWith %in% c("A", "Z")) %>%
# Make the plot
ggplot(aes(x = year, y = lifeExp, color = continent)) +
geom_line() +
facet_wrap( ~ country)
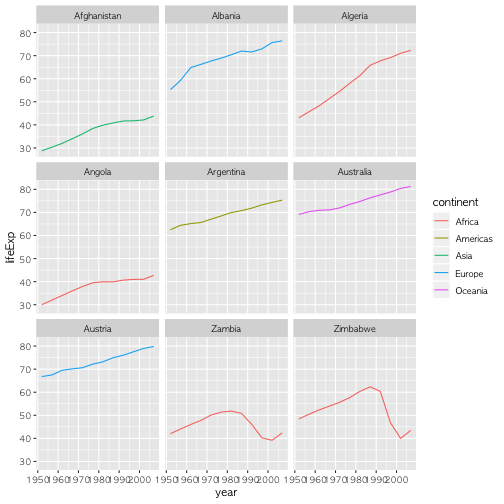
dplyr 함수를 사용하게 되면 문제를 단순화할 수 있다. 예를 들어,
첫 두단계를 다음과 같이 조합할 수도 있다:
gapminder %>%
# Filter countries that start with "A" or "Z"
filter(substr(country, start = 1, stop = 1) %in% c("A", "Z")) %>%
# Make the plot
ggplot(aes(x = year, y = lifeExp, color = continent)) +
geom_line() +
facet_wrap( ~ country)
고급 도전과제
각 대륙별로 국가를 두개 임의로 뽑아서 2002년 평균 기대수명을 계산해보자. 그리고 나서, 역순으로 대륙명을 정렬한다. 힌트:
dplyr팩키지arrange(),sample_n()함수를 사용한다. 두 함수 모두 다른dplyr함수와 유사한 구문을 갖고 있다.고급 도전과제에 대한 해답
lifeExp_2countries_bycontinents <- gapminder %>% filter(year==2002) %>% group_by(continent) %>% sample_n(2) %>% summarize(mean_lifeExp=mean(lifeExp)) %>% arrange(desc(mean_lifeExp))
추가 학습 교재
- R for Data Science
- Data Wrangling Cheat sheet
- Introduction to dplyr
- Data wrangling with R and RStudio
Key Points
데이터프레임을 조작하는데
dplyr팩키지를 사용한다.
select()함수를 사용해서 데이터프레임에서 칼럼 변수를 고를 수 있다.
filter()함수를 사용해서 행 값에 따라 데이터를 추출한다.
group_by()와summarize()함수를 사용해서 데이터 요약 작업을 수행한다.
mutate()함수를 사용해서 변수를 새로 생성한다.