R 프로그래밍
환자 데이터 분석
학습목표
- 파일에서 표 형식의 데이터를 프로그램으로 읽어들인다.
- 값을 변수에 할당한다.
- 개별적인 값과 데이터에서 일부분을 선택한다.
- 데이터에 대한 데이터프레임에 연산작업을 수행한다.
- 간단한 그래프를 화면에 출력한다.
관절염에 대한 새로운 치료법이 처방된 환자의 염증에 대한 연구를 진행하고 있고, 첫 데이터셋(Data Set) 12개를 분석할 필요가 있다. 데이터셋은 CSV 형식(comma-separated values, 구분자가 콤마 값을 가진 파일 형식)으로 저장되어 있다: 각 행은 환자 한명에 대한 정보로 구성되고, 열은 연속된 날짜 정보를 나타낸다. 첫번째 파일에 대한 처음 행 몇줄 정보는 다음과 같다:
0,0,1,3,1,2,4,7,8,3,3,3,10,5,7,4,7,7,12,18,6,13,11,11,7,7,4,6,8,8,4,4,5,7,3,4,2,3,0,0
0,1,2,1,2,1,3,2,2,6,10,11,5,9,4,4,7,16,8,6,18,4,12,5,12,7,11,5,11,3,3,5,4,4,5,5,1,1,0,1
0,1,1,3,3,2,6,2,5,9,5,7,4,5,4,15,5,11,9,10,19,14,12,17,7,12,11,7,4,2,10,5,4,2,2,3,2,2,1,1
0,0,2,0,4,2,2,1,6,7,10,7,9,13,8,8,15,10,10,7,17,4,4,7,6,15,6,4,9,11,3,5,6,3,3,4,2,3,2,1
0,1,1,3,3,1,3,5,2,4,4,7,6,5,3,10,8,10,6,17,9,14,9,7,13,9,12,6,7,7,9,6,3,2,2,4,2,0,1,1다음을 수행해야 된다.
- CSV 형식 데이터 파일을 주기억장치에 적재(loading)한다.
- 모든 환자에 대해서 각 날짜별로 평균 염증을 계산한다.
- 결과값을 도식화한다.
상기 작업을 수행하기 위해서, 프로그래밍에 관해 약간 학습할 필요가 있다.
데이터 적재하기
염증 데이터를 적재(loading)하기 위해서, 먼저 값을 담고 있는 파일이 어디에 있는지 컴퓨터에 일러줄 필요가 있다. 파일 명칭이 inflammation-01.csv라고 들었다. R에게 이것은 매우 중요하다. 만약 이 절차를 잊게되면, 파일을 읽어 올 때 오류 메시지가 나온다. 현재 작업 디렉토리를 setwd 함수를 사용해서 변경할 수 있다. 이번 예제에서, 방금전에 생성한 디렉토리로 경로를 변경한다.
setwd("~/Desktop/r-novice-inflammation/)유닉스 쉘과 마찬가지로 명령어를 타이핑하고 엔터(Enter) (혹은 리턴(return)) 키를 누른다. 다른 방식으로, RStudio GUI의 메뉴 옵션(Session -> Set Working Directory -> Choose Directory...)을 사용하여 작업 디렉토리를 변경할 수 있다.
작업 디렉토리 내부에 data 디렉토리에 데이터 파일이 위치해 있다. 이제 read.csv를 사용하여 데이터를 R로 적재할 수 있다:
read.csv(file = "data/inflammation-01.csv", header = FALSE)read.csv(...) 표현식은 함수 호출(function call)로 R에게 요청하여 read.csv 함수를 실행하게 한다.
read.csv는 두 개의 인자(arguments)가 있다: 하나는 읽고자 하는 파일 이름이고, 다른 하나는 파일 첫 행에 데이터 칼럼(열) 이름 포함 여부다. 파일이름은 문자열(string)로 될 필요가 있어서, 인용부호안에 파일이름을 넣는다. 두번째 인수 header가 FALSE로 지정된 것은 데이터 파일이 칼럼 헤더(column header)를 가지고 있지 않음을 나타낸다. FALSE 값과 반대의 경우 TRUE가 되는 것은 4번째 학습에서 더 논의를 진행할 것이다.
함수의 유용성은 무슨 값이 인자로 전달되든지 주어진 행동을 수행한다는 것이다. 예를 들어, file 인자에 다른 파일 이름을 제공하면, read.csv는 대신에 해당 파일을 읽어 들일 것이다. 다음 학습에서 함수와 인자에 관해서 좀더 자세한 내용을 배울 것이다.
함수 출력결과에 대해서 어떤 특별한 것도 지시하지 않아서, 콘솔에서 inflammation-01.csv 파일 전체 내용을 화면에 출력한다. 시도해 보세요.
read.csv는 파일을 읽어들이지만, 데이터프레임을 변수로 할당하지 않으면, 데이터를 사용할 수 없다. 변수는 x, current_temperature, subject_id처럼 단순히 값에 대한 이름이다. 새로운 변수를 생성하여 <-을 사용해서 값을 변수에 할당할 수 있다.
weight_kg <- 55변수가 값을 가지게 되면, 변수 이름을 타이핑하고 엔터(Enter) 혹은 리턴(return)을 쳐서 변수를 출력할 수 있다. 일반적으로 변수에 할당하는 경우를 제외하고 R은 함수나 연산에서 반환되는 임의의 객체를 콘솔에 출력한다.
weight_kg[1] 55
변수로 간단한 산수를 할 수 있다:
# weight in pounds:
2.2 * weight_kg[1] 121
새로운 값을 할당함으로써 객체의 값을 변경할 수 있다:
weight_kg <- 57.5
# 체중이 이제 킬로그램 단위다.
weight_kg[1] 57.5만약 변수를 이름이 써진 포스트잇 같은 스티커 노트라고 가정한다면, 할당은 특정한 값에 스티커 노트를 붙이는 것과 같다:
이것이 의미하는 바는 한 객체에 값을 할당하는 것이 다른 변수의 값을 변경시키지는 않는다. 예를 들어, 변수에 특정 개체에 대한 무게를 파운드로 저장하자:
weight_lb <- 2.2 * weight_kg
# kg 단위 체중...
weight_kg[1] 57.5# ...그리고 파운드 단위 체중
weight_lb[1] 126.5그리고 나서 weight_kg를 변경하자:
weight_kg <- 100.0
# 이제 kg 단위 체중...
weight_kg[1] 100# ...파운드 체중은 그대로
weight_lb[1] 126.5weight_lb 변수는 값이 어디에서 왔는지 기억하지 않기 때문에, 자동적으로 weight_kg이 변경될 때 갱신되지 않는다. 엑셀같은 스프레드쉬트가 동작하는 방식과 이런 점이 다르다.
변수에 어떻게 값을 할당하는지 알기 때문에, read.csv를 다시 실행하고 결과를 저장하자:
dat <- read.csv(file = "data/inflammation-01.csv", header = FALSE)상기 문장은 어떤 출력결과도 만들어 내지 않는데 이유는 할당은 어떤 것도 화면에 출력하지 않기 때문이다. 데이터가 적재되었는지 확인하고자 한다면, 변수값을 출력할 수 있다. 하지만, 매우 큰 데이터셋에 대해서 데이터 첫 몇 줄만 화면에 출력하는 함수 head를 사용하는 것이 편리하고 좋다.
head(dat) V1 V2 V3 V4 V5 V6 V7 V8 V9 V10 V11 V12 V13 V14 V15 V16 V17 V18 V19 V20
1 0 0 1 3 1 2 4 7 8 3 3 3 10 5 7 4 7 7 12 18
2 0 1 2 1 2 1 3 2 2 6 10 11 5 9 4 4 7 16 8 6
3 0 1 1 3 3 2 6 2 5 9 5 7 4 5 4 15 5 11 9 10
4 0 0 2 0 4 2 2 1 6 7 10 7 9 13 8 8 15 10 10 7
5 0 1 1 3 3 1 3 5 2 4 4 7 6 5 3 10 8 10 6 17
6 0 0 1 2 2 4 2 1 6 4 7 6 6 9 9 15 4 16 18 12
V21 V22 V23 V24 V25 V26 V27 V28 V29 V30 V31 V32 V33 V34 V35 V36 V37 V38
1 6 13 11 11 7 7 4 6 8 8 4 4 5 7 3 4 2 3
2 18 4 12 5 12 7 11 5 11 3 3 5 4 4 5 5 1 1
3 19 14 12 17 7 12 11 7 4 2 10 5 4 2 2 3 2 2
4 17 4 4 7 6 15 6 4 9 11 3 5 6 3 3 4 2 3
5 9 14 9 7 13 9 12 6 7 7 9 6 3 2 2 4 2 0
6 12 5 18 9 5 3 10 3 12 7 8 4 7 3 5 4 4 3
V39 V40
1 0 0
2 0 1
3 1 1
4 2 1
5 1 1
6 2 1
도전과제 - 변수에 값을 할당한다
도표를 그려서, 다음 프로그램의 각 문장이 실행된 뒤에, 어떤 변수가 어떤 값을 참조하는지 보이세요:
mass <- 47.5
age <- 122
mass <- mass * 2.0
age <- age - 20데이터 능숙하게 다루기
데이터가 주기억장치에 올라갔기 때문에, 데이터를 가지고 무언가를 시작할 수 있다. 먼저 dat가 무슨 형식인지 확인해보자:
class(dat)[1] "data.frame"출력결과를 통해서 현재 데이터가 데이터프레임(data frame)이라는 것을 알 수 있다. 많은 사람들이 사용해서 익숙한 마이크로소프트 엑셀 스프레드쉬트와 유사하다. 데이터프레임이 데이터를 저장하는데 매우 유용하고, R로 프로그래밍하는 곳 어디서나 발견하게 된다. 실험 데이터에 대한 전형적인 데이터프레임은 행에는 개별 관측점, 열에는 변수가 담겨진다.
함수 dim으로 데이터프레임에 대한 형태(shape), 즉, 차원(dimensions)을 볼 수 있다:
dim(dat)[1] 60 40출력결과를 통해서 dat 데이터프레임이 60 행과 40 열로 구성된 것을 알 수 있다.
데이터프레임에서 값 하나를 얻으려고 한다면, 수학에서 하는 것과 같은 방식으로 꺾쇄 괄호내부에 인덱스(index)를 넣을 수 있다:
# dat 첫번째 값
dat[1, 1][1] 0# dat 중간값
dat[30, 20][1] 16[30, 20] 처럼 인텍스를 넣어서 데이터프레임의 요소값을 하나 선택할 수 있지만, 전체 부문도 선택할 수 있다. 예를 들어, 다음과 같이 첫 환자 네명(행)에 대해서, 첫 10일치(열) 값을 선택할 수 있다:
dat[1:4, 1:10] V1 V2 V3 V4 V5 V6 V7 V8 V9 V10
1 0 0 1 3 1 2 4 7 8 3
2 0 1 2 1 2 1 3 2 2 6
3 0 1 1 3 3 2 6 2 5 9
4 0 0 2 0 4 2 2 1 6 7슬라이스(slice) 1:4가 뜻하는 것은 “1번 인덱스에서 시작해서 4번 인덱스까지다.”
슬라이스가 반드시 1번에서 시작할 필요는 없다. 즉, 아래 행은 5~10번 행을 선택한 사례다:
dat[5:10, 1:10] V1 V2 V3 V4 V5 V6 V7 V8 V9 V10
5 0 1 1 3 3 1 3 5 2 4
6 0 0 1 2 2 4 2 1 6 4
7 0 0 2 2 4 2 2 5 5 8
8 0 0 1 2 3 1 2 3 5 3
9 0 0 0 3 1 5 6 5 5 8
10 0 1 1 2 1 3 5 3 5 8c 함수(combine을 뜻함)를 사용해서 인접하지 않는 값도 선택할 수 있다:
dat[c(3, 8, 37, 56), c(10, 14, 29)] V10 V14 V29
3 9 5 4
8 3 5 6
37 6 9 10
56 7 11 9또한, 행과 열에 슬라이스를 줄 필요도 없다. 만약 행에 슬라이스가 없다면, R은 모든 행을 반환한다. 만약 열에 슬라이스가 없다면, R은 모든 열을 반환한다. dat[, ]처럼 행과 열 모두에 슬라이가 없다면 R은 전체 데이터프레임을 반환한다.
# 5번째 행에 대한 모든 열
dat[5, ] V1 V2 V3 V4 V5 V6 V7 V8 V9 V10 V11 V12 V13 V14 V15 V16 V17 V18 V19 V20
5 0 1 1 3 3 1 3 5 2 4 4 7 6 5 3 10 8 10 6 17
V21 V22 V23 V24 V25 V26 V27 V28 V29 V30 V31 V32 V33 V34 V35 V36 V37 V38
5 9 14 9 7 13 9 12 6 7 7 9 6 3 2 2 4 2 0
V39 V40
5 1 1# 16번째 열에 대한 모든 행
dat[, 16] [1] 4 4 15 8 10 15 13 9 11 6 3 8 12 3 5 10 11 4 11 13 15 5 14
[24] 13 4 9 13 6 7 6 14 3 15 4 15 11 7 10 15 6 5 6 15 11 15 6
[47] 11 15 14 4 10 15 11 6 13 8 4 13 12 9이제 염증데이터에 대해서 좀더 살펴보는데, 일반적인 수학 연산을 수행해보자. 데이터를 분석할 때, 환자마다 최대값 혹은 날마다 평균값 같은 통계량을 살펴보고자 한다. 이것을 수행하는 방법은 새로이 임시 데이터프레임을 생성하고 나서 데이터를 선택하고, 해당 개체에 대한 연산작업을 수행하는 것이다:
# 첫번째 행과, 모든 열
patient_1 <- dat[1, ]
# 1번 환자에 대한 최대 염증값
max(patient_1)[1] 18사실 변수에 행을 저장할 필요는 없다. 대신에, 데이터 선택과 함수 호출을 조합할 수 있다:
# 2번 환자에 대한 최대 염증값
max(dat[2, ])[1] 18R은 또한 데이터에 대한 최소값, 평균, 중위값, 표준편차 같은 일반적인 연산작업에 대한 함수를 제공한다:
# 7번째 날 최소 염증값
min(dat[, 7])[1] 1# 7번째 날 평균 염증값
mean(dat[, 7])[1] 3.8# 7번째 날 염증값 중위수
median(dat[, 7])[1] 4# 7번째 날 염증값 표준편차
sd(dat[, 7])[1] 1.725187만약 모든 환자에 대한 최대 염증값 혹은 각 날짜별로 평균값이 필요하다면 어떨까? 다음 도표처럼, 데이터프레임의 가장자리를 따라서 연산을 수행하고자 한다:
상기 연산을 수행하기 위해서, apply 함수를 사용한다.
apply 함수를 사용해서 데이터프레임의 모든 행(MARGIN = 1) 혹은 열(MARGIN = 2)에 동일한 함수연산을 수행한다.
그래서, 각 환자별로 평균 염증값을 얻기 위해서, 데이터프레임의 모든 행(MARGIN = 1)에 대해 평균값을 계산할 필요가 있다.
avg_patient_inflammation <- apply(dat, 1, mean)각 날짜별 평균 염증값을 얻기 위해서, 데이터프레임의 모든 열(MARGIN = 2)에 대해 평균값을 계산할 필요가 있을 것이다.
avg_day_inflammation <- apply(dat, 2, mean)apply 함수의 두번째 인자는 MARGIN이여서, 상기 명령어는 apply(dat, MARGIN = 2, mean)과 동일하다.
도전과제 - 데이터 슬라이싱(부분집합, subsetting)
데이터프레임 부분집합(subset)를 슬라이스(slice)라고 한다. 문자 벡터에도 슬라이스를 적용할 수 있다:
animal <- c("m", "o", "n", "k", "e", "y")
# 첫 문자 세개
animal[1:3][1] "m" "o" "n"# 마지막 문자 세개
animal[4:6][1] "k" "e" "y"animal[1:4]슬라이스를 사용해서 첫 네개 문자를 선택했다면, 역순으로 첫 네개 문자를 어떻게 얻을 수 있을까?animal[-1]의 값은 무엇일까?animal[-4]의 값은 무엇일까? 상기 해답이 주어졌을 때,animal[-1:-4]은 무엇이 될지 설명해 보세요.animal슬라이스를 사용해서 단어 “eon”을 예를 들어c("e", "o", "n")처럼 작성하는 새로운 문자 벡터를 생성하세요.
도전과제 - 데이터 부분집합 2
5번째 환자에 대해서 3일에서 7일에 걸친 최대 염증값을 알아내고자 한다고 가정하자. 이 작업을 위해서, 데이터프레임으로부터 연관된 슬라이스를 뽑아내고 최대값을 계산한다. 다음 R 코드 중 어떤 것이 올바른 정답을 제시하나요?
max(dat[5, ])max(dat[3:7, 5])max(dat[5, 3:7])max(dat[5, 3, 7])
도식화(Plotting)
수학자 Richard Hamming은 “컴퓨팅의 목적은 숫자가 아니라 직관(insight)이다.”라고 말했다. 그래서 직관을 키우는 가장 좋은 방법은 흔히 데이터를 시각화하는 것이다. 시각화에 대해 그 자체로 전체 강의를 펼칠만 하지만, 여기서는 R의 도식화 기능 몇가지만 살펴본다.
시간에 따른 평균 염증값을 살펴보자. apply(dat, 2, mean)을 사용해서 이미 값을 계산해서, 변수 avg_day_inflammation에 저장했다. 이를 그래프로 도식화하는 것을 plot 함수로 할 수 있다:
plot(avg_day_inflammation)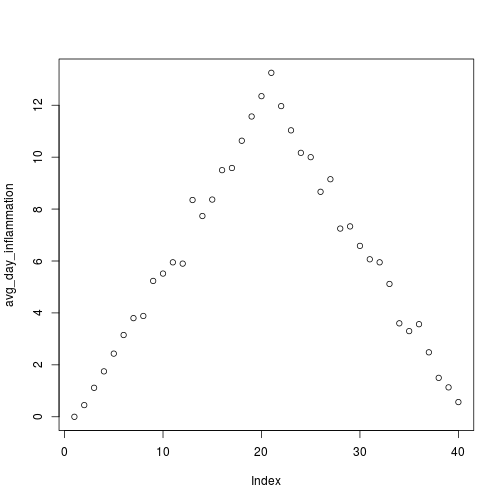
위에서 plot 함수에 모든 환자에 대한 일별 평균 염증값에 대응되는 숫자 벡터를 전달했다. plot 함수는 Y축에 평균 염증 수준과 X축에 이 경우 40일 처방에 해당하는 벡터값의 순서, 즉 인덱스를 두고 산점도(scatter plot)를 생성했다. 결과는 대략 선형적으로 올라가고 내려가지만 의심스럽다. 다른 연구 결과에 따르면, 좀더 빠른 급격한 상승과 좀더 완만한 하강이 예측됐다. 또 다른 통계량 두개(일자별 최대 그리고 최소 염증값)을 살펴보자.
max_day_inflammation <- apply(dat, 2, max)
plot(max_day_inflammation)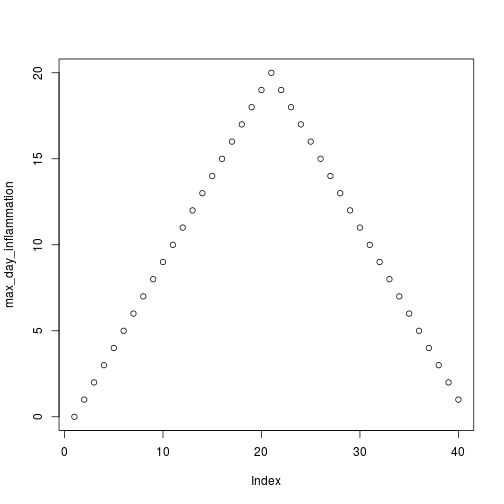
min_day_inflammation <- apply(dat, 2, min)
plot(min_day_inflammation)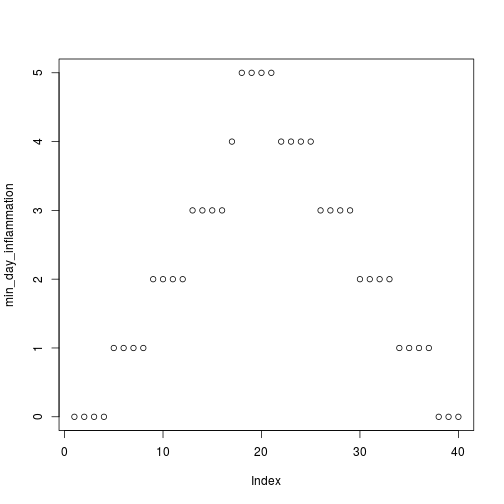
최소값은 계단 함수(Step function) 모양으로 보이지만, 최대값은 완벽하게 매끄럽게 상승하고 하강하고 있다. 어느쪽의 결과도 그럴듯하게 보이지 않아서, 계산에서 실수가 있거나 데이터에 무언가 잘못된 것이 있다.
도전과제 - 데이터 도식화
모든 환자에 대해서 각 일자별로 염증 데이터의 표준편차를 보이는 그래프를 생성하세요.
